Med spatial transkriptomik är det möjligt att kartlägga vilka gener som uttrycks i vilka celler i en vävnadsbit. Med hjälp av informationen om varje enskild cells genaktivitet kan forskarna börja analysera hur celler pratar med varandra och hur detta påverkas av viruset.
Texten är en fördjupning till tidningsartikeln ”Så går snacket vid en virusattack”.
Det så kallade centrala dogmat innebär att den genetiska informationen överförs från DNA via RNA till proteiner. Man skulle kunna likna det vid att DNA är originalritningen för proteiner, RNA är arbetskopian som används vid konstruktionen, och proteinerna är slutprodukten som har många olika funktioner. Som byggmateriel i kroppen (bygger t.ex. upp muskler) eller som arbetare som verkställer olika funktioner (t.ex. enzymer som bryter ner maten).
mRNA (messenger RNA; budbärar-RNA på svenska) är mer eller mindre långa trådlika molekyler uppbyggda av nukleotider, molekylära byggstenar som nukleinsyrorna består av. mRNA-innehållet i en cell vid en given tidpunkt avspeglar vilka gener som är uttrycks, det vill säga är aktiva för stunden.
Så går det till
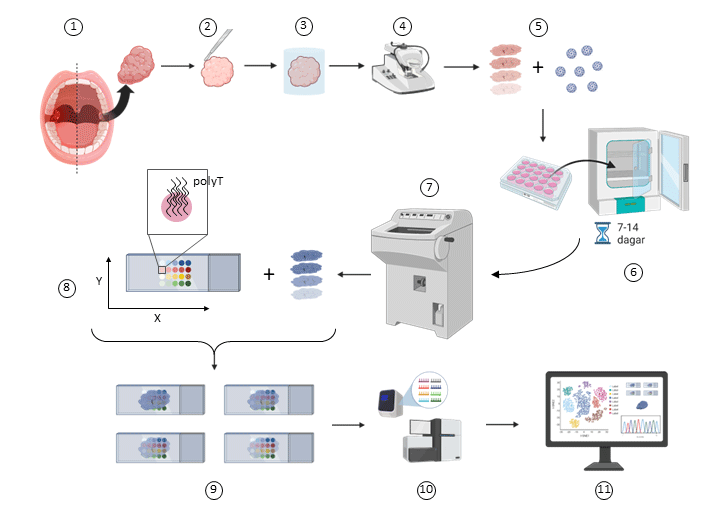
2. Dissekering av halsmandelvävnad och borttagning av död vävnad.
3. Inbäddning av halsmandelvävnad i agarosgel för att möjliggöra mikrotomsnittning av levande halsmandelvävnad.
4. Mikrotomsnittning (snitt: 300-500 mikrometer).
5. Placering av vävnadssnitt på små flottar i odlingsplatta. Virus tillsätts sedan för infektion av halsmandelvävnad.
6. Infektionsförloppet följs sedan i upp till två veckor för att möjliggöra kartläggning av hur virus etablerar infektion och hur vårt initiala immunförsvar reagerar på infektionen.
7. De infekterade vävnadssnitten fryses sedan ner i ett frysmedium, för att sedan snittas i mycket tunna snitt i en så kallad cryostat (10 mikrometer).
8. Snitten placeras sedan på ett glas fullt med polyT-svansar organiserade i små cirkulära kluster. Varje cirkulärt kluster av polyT-svansar har specifika X- och Y-koordinater. Detta gör det möjligt att fånga upp varje enskild cells genuttryck i dess rätta position i vävnaden.
9. Genom att analysera flera snitt från samma vävnadsbit kan en tredimensionell kartläggning av genuttrycket i halsmandelvävnaden rekonstrueras.
10. De uppfångade mRNA-transkripten sekvenseras sedan för att generera den data som sedan analyseras.
11. Producerad data analyseras med bioinformatik och statistik för att kartlägga genuttrycket i vävnaden. Detta gör det möjligt att bestämma vilka celler som aktiveras av infektionen och hur de aktiveras, interagerar och signalerar till varandra för att generarea ett immunologiskt svar mot virusinfektionen.
När forskarna vill studera exempelvis ett infektionsförlopp utgår de från tunna vävnadsbitar av tonsiller som placeras på små ”flottar” i en näringslösning. Vävnadsbitarna infekteras med virus och forskarna följer förloppet genom att samla vävnadsprover vid olika tidpunkter efter infektion (1-6).
Varje provtagen vävnadsbit fryses ner och skivas upp i mycket tunna snitt som motsvarar ungefär en cells tjocklek. Samtidigt förbereds en så kallad ”capture area”, en fångstyta. Det är en del av en glasskiva (6,5×6,5 mm) som täckts med många små ”prickar” där varje prick består av en mängd korta polyT-strängar (se nedanstående stycke) som fästs på glasskivan. Varje prick är märkt med specifika x/y-koordinater vilket gör det möjligt att placera varje enskild cells genutryck på dess rätta plats i vävnaden (7-8).
Alla mRNA har en så kallad polyA-svans i slutet av molekylen. Med polyA menas att svansen är uppbyggd av enbart nukleotiden adenin (A). PolyA-svansarna är enkelsträngade och parar gärna ihop sig med polyT-svansar, något som utnyttjas i den här tekniken.
De supertunna vävnadsbitarna läggs nu över fångstytan och eftersom varje prick motsvarar ungefär en cells storlek kommer varje prick att fånga upp mRNA-innehållet i en enskild cell. Detta kan sedan upprepas med flera uppskivade supertunna vävnadsbitar för att skapa en tredimensionell bild av hur genuttrycket ser ut i vävanden (forts. 7-8).
mRNA-innehållet i varje prick avspeglar genaktiviteten hos cellen som var placerad över just den pricken. Koordinatsystemet gör det möjligt för forskarna att identifiera varje cells placering i den undersökta vävnadsbiten. Med hjälp av informationen om varje enskild cells genaktivitet kan forskarna börja analysera hur celler pratar med varandra och hur detta påverkas av viruset (9-11).
Tekniken har utvecklats av forskare vid Kungliga tekniska högskolan, KTH och Karolinska institutet, KI.
Text: EVA BARTONEK ROXÅ
Illustration, bildtext och faktagranskning: JOAKIM ESBJÖRNSSON-KLEMENDZ